
 |
|||||
|
|
|||||
|
IDENTIFIZIERUNG PAK (POLYCYKLISCHE AROMATISCHE KOHLENWASSERSTOFFE) ABBAUENDER BODENBAKTERIEN MITTELS GENTECHNISCHER NACHWEISMETHODEN |
|
|
||||||||||||||||||||||||||||||||||||||||||||||
|
Problem: |
|
Strategie: |
|
|
||||||||||||||||||||||||||||||||||||||||
|
Folgerung: |
|
|
||||||||||||||||||||||||||||||||||||||||
|
Folgerung: |
|
|
||||||||||||||||||||||||||||||
|
Detektion ndoB-homologer Gene in verschiedenen PAK-abbauenden Reinkulturen durch Southern-Blot- Hybridisierungen. |
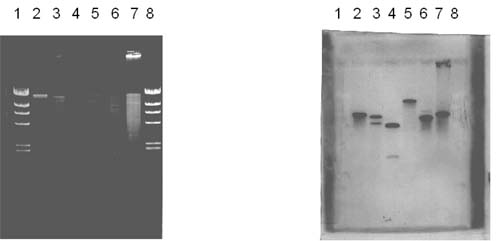 |
|
|
|
Schlußfolgerung: Die dargestellten Methoden der Gensonden- und PCR-Technik ermöglichen eine aussagekräftige Dar- stellung des PAK- Abbaupotentials von Bakterienpopulationen und damit eine verbesserte Prozesskontrolle in biologischen Dekontaminations- verfahren (Monitoring der Populationsentwicklung; gezieltes Erstellen von schadstoffspezifischen Animpfkulturen und Verfolgung des Schicksals entsprechender Kulturen während des Sanierungsprozesses). |
|
|